Paper Details
Title: V-Net: Fully Convolutional Neural Networks for Volumetric Medical Image Segmentation
Authors: Fausto Milletari, Nassir Navab, Seyed-Ahmad Ahmadi
Conference: Medical Image Computing and Computer-Assisted Intervention (MICCAI)
Year of Publication: 2016
Link: https://arxiv.org/abs/1606.04797
Key Focus: This paper introduces V-Net, a fully convolutional neural network designed for 3D volumetric medical image segmentation. It processes MRI volumes end-to-end, leveraging volumetric convolutions instead of slice-wise approaches. V-Net adopts a novel loss function based on the Dice coefficient to handle class imbalance between foreground and background voxels, avoiding the need for re-weighted loss functions. The architecture includes a symmetric compression-decompression design with residual learning in each stage to ensure fast convergence and high accuracy. Data augmentation techniques, such as random deformations and histogram matching, enhance training performance on limited datasets. V-Net achieves state-of-the-art results on the PROMISE 2012 dataset for prostate MRI segmentation, providing accurate segmentations in under a second on modern GPUs.
Comparison to U-Net: While U-Net focuses on 2D biomedical image segmentation, V-Net is tailored for 3D volumetric segmentation, making it more suitable for tasks involving MRI and other volumetric data. V-Net's Dice-based loss function and residual learning contribute to its robust handling of imbalanced medical datasets and faster convergence.
💡Key point
📄 Summary of the Paper
1. Introduction
초기 CNN 기반 의료 이미지 세그먼테이션은 패치 단위로 국소적 정보를 분류했으나, 이는 국소적 맥락만 고려하여 오류가 발생하기 쉽다는 단점이 있었다. 특히 초음파 같은 복잡한 데이터는 잘못 분류된 영역이 많아지기도 했다. 최근 연구는 CNN 예측을 마코프 랜덤 필드나 투표 전략, 레벨 셋과 같은 방법과 결합하는 방식으로 발전했지만, 패치 단위 접근법은 계산 비용이 높다는 한계가 있다.
기존의 완전 합성곱 네트워크(FCN)는 2D 이미지에만 적용되었으며, 이러한 모델은 RGB 이미지 또는 현미경 데이터를 대상으로 하였다. 하지만 본 연구에서는 기존 방식과 달리 입력 볼륨을 슬라이스 단위로 처리하지 않고 3D 볼륨 합성곱을 활용하며, Dice 계수를 기반으로 한 손실 함수를 제안해 훈련 중 최적화를 수행하고자 한다. 제안된 방법은 전립선 MRI 테스트 데이터에서 빠르고 정확한 성능을 보여주었으며, 동일한 테스트 데이터로 평가된 기존 방법과 직접 비교를 수행하였다.

2. Method

네트워크의 왼쪽 부분이 데이터를 압축하는 압축 경로, 오른쪽이 신호를 원래 크기로 복원하는 복원 경로인 것은 U-Net과 동일하다!
각 부분에 대핸 상세 구조와 특징은 다음과 같다.
- 압축 경로(Compression Path):
- 데이터의 해상도를 줄이면서 특징을 추출함.
- 각 단계는 5×5×5 볼륨 커널과 스트라이드 2를 사용하여 해상도를 절반으로 줄임.
- 잔차 학습(residual learning)을 통해 빠른 수렴을 보장함.
- 복원 경로(Decompression Path):
- 압축 과정에서 손실될 수 있는 세부 정보를 복구함.
- 디컨볼루션 연산을 통해 데이터 해상도를 복원하며 전경과 배경을 구분하는 두 개의 채널 맵을 생성함.

풀링 대신 합성곱 연산은 사용하는데, 이는 메모리 효율성 증가와 네트워크에 대한 더 나은 해석 가능성 제공이라는 측면에서 장점을 갖는다. 각 단계에서 특징 채널 수를 두 배로 증가시켜서 더욱 풍부한 특징을 추출한다.
또한, 초기 단계의 정보를 재사용하는데, 압축 경로에서 추출된 초기 정보를 복원 경로로 전달하여 세부 정보를 유지한다. 이는 경계 품질을 높이면서도 학습속도를 단축시킨다는 장점을 갖는다.

위의 Table 1은 V-Net의 각 합성곱 레이어 단계에서 입력 크기와 수용 영역(Receptive Field)을 나타낸 것이다. 수용 영역은 네트워크가 해당 레이어에서 고려하는 입력 데이터의 공간 범위를 나타내며, 네트워크가 깊어질수록 수용 영역이 확장되어 전체 볼륨의 전역 정보를 학습할 수 있음을 보여준다.
3. Dice loss layer
기존의 접근법들은 학습 중에 전경 영역에 배경 영역보다 더 높은 중요도를 부여하기 위해 표본 재가중치(sample re-weighting)를 기반으로 한 손실 함수를 사용하였다. 이는 전경이 스캔의 작은 영역만 차지하여 배경에 편향된 학습이 발생한다는 문제점이 있었고, 전경에 가중치를 부여하는 표본 재가중치 방법을 사용하더라도 설정과 구현이 복잡하다는 단점이 있었다. 따라서 본 연구에서는 Dice 계수를 기반으로 한 새로운 목적 함수를 제안하였다.
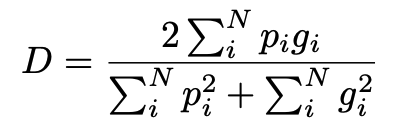
Dice 계수는 0과 1 사이의 값을 가지며, 이를 최대화하는 것을 목표로 한다.
여기서 합은 예측된 이진 세그먼테이션 볼륨 $P$의 predicted binary segmentation volume $p_i$와 ground truth binary volume $g_i$에 의해 계산된다.
위의 Dice 함수를 미분한 결과는 다음과 같다.

위의 기울기는 예측된 결과(prediction)의 j-th voxel에 대해 계산된다.
위의 공식을 사용하면 클래스 간 데이터 비율을 균형 맞추기 위해 별도의 가중치를 할당하지 않아도 되며, 동시에 실험적으로 동일한 네트워크를 다중항 로지스틱 손실 함수와 표본 재가중치를 사용해 최적화한 경우보다 훨씬 우수한 결과를 얻을 수 있다.
3.1. Training
학습 과정에서 전립선 MRI 스캔 데이터를 사용하며, 모든 볼륨은 128 × 128 × 64 보oxel 크기와 1 × 1 × 1.5mm 공간 해상도를 가진다.
의료 데이터는 전문가의 수작업 주석이 필요하므로 수집 비용이 높고 데이터 양이 제한적이라는 단점이 있다.
따라서 다음과 같은 방법으로 데이터를 증강한다.
- 랜덤 변형: 2 × 2 × 2 크기의 제어점을 활용하고 B-스플라인 보간법을 사용하여 밀도 변형 필드를 생성한다.
- 히스토그램 매칭: 학습 볼륨의 강도 분포를 데이터셋 내 다른 랜덤 스캔과 일치시켜 다양성을 증가시킨다.
이러한 증강은 "실시간(on-the-fly)"으로 수행되어 저장 공간 문제를 최소화한다.
3.2. Testing
이전에 보지 못한 MRI 볼륨은 네트워크를 통해 순방향(feed-forward) 방식으로 처리하여 세그먼테이션이 가능하다. 마지막 합성곱 레이어 출력은 소프트맥스 연산 이후 배경과 전경에 대한 확률 맵으로 구성된다. 전경에 속할 확률이 배경보다 높은 voxel(> 0.5)은 해부학적 구조의 일부로 간주된다고 한다.
4. Results
실험 결과는 다음과 같다.
(본 포스팅에서는 간단히 설명하도록 하겠다.)
PROMISE 2012 데이터셋 기반 결과

- V-Net은 PROMISE 2012 챌린지 데이터셋에서 훈련됨.
- 50개의 MRI 볼륨과 해당하는 수동 주석 데이터를 사용해 훈련을 진행함.
- 이 데이터셋은 다양한 병원, 장비, 획득 프로토콜에서 수집된 의료 데이터를 포함하여 임상 환경의 다양성과 도전 과제를 대표함.

- 그래프는 테스트 데이터에서 세그먼트된 MRI 볼륨들이 각 Dice 계수 구간에 얼마나 분포했는지를 보여주며, 각 Dice 계수 구간(bin)에 해당하는 볼륨의 개수를 막대 그래프로 표현하였다.
- 다른 방법(예: ScrAutoProstate, Grislies 등)과 V-Net의 Dice 기반 손실(Dice-based loss)을 비교한 결과이다.
- V-Net의 Dice 기반 손실은 0.8 이상의 높은 Dice 계수를 가진 볼륨이 대부분을 차지하며, 다른 방법에 비해 높은 성능을 일관되게 유지하며, 0.9 이상에서도 상당히 많은 볼륨이 분포함을 볼 수 있다.

- V-Net (Dice 기반 손실):
- 평균 Dice 계수: 0.869 ± 0.033
- 평균 Hausdorff 거리: 5.71 ± 1.20mm
- 챌린지 점수: 82.39
- 다른 알고리즘과 비교 시, Dice 계수와 Hausdorff 거리 모두 우수한 성능을 기록하였다.
- 소프트맥스 기반 손실 함수(V-Net + mult. logistic loss) 대비 약 12% 높은 챌린지 점수와 우수한 거리 감소를 보여준다.

- 두 손실 함수(Dice Loss vs. Re-weighted Soft-Max Loss)를 사용해 세그먼트된 MRI 결과를 시각적으로 비교한 그림으로 초록색(Dice Loss)과 노란색(Soft-Max Loss)으로 예측된 영역을 비교하고 있다.
- Dice Loss를 사용한 경우 해부학적 구조를 더 정확히 캡처하며, 전경과 배경의 경계가 더 정밀하며, 소프트맥스 손실은 배경과 경계 부분에서 누락되거나 부정확한 세그먼테이션을 보인다.
5. Conclusion
본 연구는 전립선 MRI 볼륨을 빠르고 정확하게 세그먼테이션할 수 있는 볼륨 합성곱 신경망(Volumetric Convolutional Neural Network)을 기반으로 한 접근 방식을 제시하고 있다. 특히 Dice 중첩 계수(Dice overlap coefficient)를 기반으로 한 새로운 목적 함수를 학습 중 최적화하여, 예측된 세그먼테이션과 실제 정답 주석 간의 유사성을 측정한다는 점에서 차별성을 갖는다. Dice 손실 계층은 배경과 전경 픽셀의 양이 매우 불균형한 경우에도 샘플 재가중치(sample re-weighting)를 필요로 하지 않으며, 이진 세그먼테이션 작업에 적합하다.
향후 연구에서는 여러 GPU에 네트워크를 분산시키는 방식을 통해 더 높은 해상도 데이터와 초음파와 같은 다른 모달리티에서 다중 영역을 포함하는 볼륨의 세그먼테이션을 목표로 할 수 있음을 시사한다.
'논문 리뷰 > Computer Vision' 카테고리의 다른 글
| [논문 리뷰] U-Net: Convolution Networks for Biomedical Image Segmentation (0) | 2024.11.18 |
|---|---|
| [논문 리뷰] AN IMAGE IS WORTH 16X16 WORDS: TRANSFORMERS FOR IMAGE RECOGNITION AT SCALE (5) | 2024.10.14 |

